RNA structure and function prediction
ผศ.ดร.ธีรศักดิ์ เอโกบล
อัพเดท 7 ตุลาคม 2663
การวิเคราะห์ชีวสารสนเทศของอาร์เอ็นเอมีความสำคัญต่อการศึกษาทางชีววิทยาในปัจจุบันเป็นอย่างยิ่ง บทเรียนนี้จะแสดงให้เห็นถึงการนำความรู้พื้นฐานเกี่ยวกับโครงสร้างและหน้าที่ของอาร์เอ็นเอมาพัฒนาเครื่องมือทางชีวสารสนเทศเพื่อตอบคำถามเกี่ยวกับการทำงาน การควบคุมและความสัมพันธ์ทางวิวัฒนาการของอาร์เอ็นเอชนิดต่างๆ ในสิ่งมีชีวิต การศึกษาทรานสคริปโตม (transcriptome) หรืออาร์เอ็นเอทั้งหมดในระบบของสิ่งมีชีวิตที่สนใจด้วยเทคนิคการ next generation sequencing หรือ RNA sequencing ทำให้ทราบถึงอาร์เอ็นเอทุกชนิดหรือเฉพาะกลุ่มที่สังเคราะห์ขึ้นในระบบที่สนใจ การนำโปรแกรมทางชีวสารสนเทศมาทำนายโครงสร้างและหน้าที่ของอาร์เอ็นเอที่ได้มาเหล่านี้จะทำให้เข้าใจกลไกระดับโมเลกุลที่เกี่ยวข้องได้ดีมากยิ่งขึ้น
1. องค์ประกอบและโครงสร้างของอาร์เอ็นเอ
อาร์เอ็นเอเป็นโมเลกุลที่มีโครงสร้างและหน้าที่ที่หลากหลาย ได้แก่ ไรโบโซมอลอาร์เอ็นเอ (ribosomal RNA หรือ rRNA) ที่เป็นองค์ประกอบของไรโบโซม เมสเซนเจอร์อาร์เอ็นเอ (messenger RNA หรือ mRNA) ที่ถ่ายทอดคำสั่งทางพันธุกรรมผ่านกระบวนการถอดรหัส (transcription) เพื่อการแปลรหัส (translation) เป็นลำดับกรดอะมิโนของโปรตีน ทรานสเฟอร์อาร์เอ็นเอ (transfer RNA หรือ tRNA) ที่มีบทบาทสำคัญในการสังเคราะห์โปรตีน และนอนโคดดิ้งอาร์เอ็นเอ (non-coding RNA หรือ ncRNAs) ซึ่งไม่ผ่านกระบวนการแปลรหัสเป็นโปรตีนแต่มีหน้าที่และบทบาทสำคัญในกระบวนการควบคุุมการทำงานของการถอดและแปลรหัสพันธุกรรม และกระบวนการระดับโมเลกุลอื่นๆ ให้เกิดขึ้นในอัตราและเวลาที่เหมาะสม
อาร์เอ็นเอสามารถแบ่งประเภทได้หลายกลุุ่ม ได้แก่ coding RNA หรือ mRNA และ non-coding RNA ซึ่งแบ่งย่อยได้อีกหลายชนิด เช่น อาร์เอ็นเอที่เกี่ยวข้องกับกระบวนการแปลรหัส (rRNA และ tRNA) short non-coding RNA (siRNA miRNA และ piRNA) และ long non-coding RNA (Santosh et al., 2015) หากอ้างอิงจากฐานข้อมูล Rfam (http://rfam.xfam.org/) ในภาพที่ 1 จะพบว่าปัจจุบันมีฐานข้อมูลนี้แบ่งอาร์เอ็นเอออกเป็น 3,446 แฟมิลี่ ได้แก่ snRNA snoRNA sRNA lncRNA CRISPR riboswitch IRES ribozyme scaRNA antitoxin และ intron เป็นต้น ฐานข้อมูลนี้ยังแบ่งข้อมูลอาร์เอ็นเอตามชนิดของสิ่งมีชีวิตไว้อีกด้วย
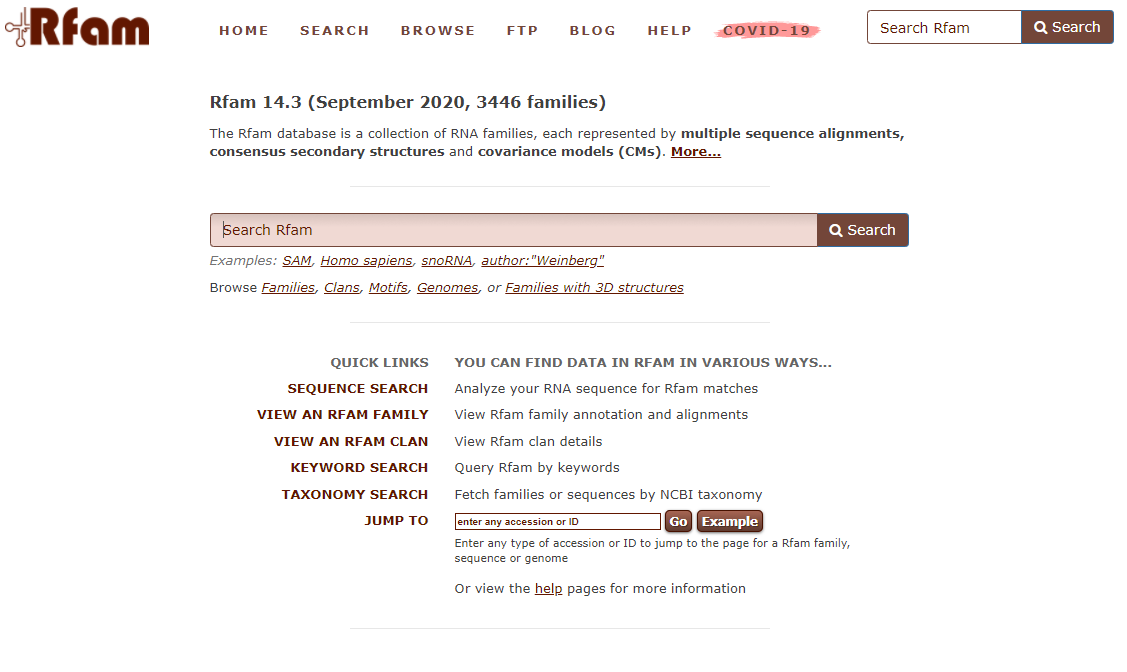
ภาพที่ 1 ฐานข้อมูล Rfam
เมื่อเปรียบเทียบความแตกต่างระหว่างอาร์เอ็นเอกับดีเอ็นเอ นอกจากจะมีชนิดของน้ำตาลเพสโตส (น้ำตาลไรโบสในอาร์เอ็นเอและน้ำตาลดีออกซีไรโบสในดีเอ็นเอ) และไนโตรจีนัสเบสหนึ่งชนิดที่แตกต่างกันคือ ยูเรซิล (uracil, U) ในอาร์เอ็นเอและไธมีน (thymine, T) ในดีเอ็นเอ อีกประเด็นสำคัญคือการเข้าคู่ของไนโตรจีนัสเบสที่จำกัดสำหรับดีเอ็นเอเพื่อการเกิดโครงสร้างของดีเอ็นเอที่เป็นเกลียวคู่คล้ายบันไดเวียน (double helix) โดยเป็นการเข้าคู่ระหว่างเบสอะดีนีน (adenine, A) และไธมีนด้วยพันธะไฮโดรเจนสองพันธะ และการเข้าคู่ระหว่างเบสกวานีน (guanine, G) และไซโทซีน (cytocine, C) ด้วยสามพันธะไฮโดรเจน ซึ่งพันธะไฮโดรเจนเหล่านี้เกิดระหว่างโมเลกุลของไนโตรจีนัสเบสที่มาเข้าคู่ ตามแบบแผนของวัตสันและคริก (Watson-Crick basepairing) แต่ในกรณีของอาร์เอ็นเอที่เป็นสายเดี่ยว (single strand) นั้นทำให้โมเลกุลมีความยืดหยุ่นในการหมุนพับโมเลกุลทำให้สามารถเกิดพันธะไฮโดรเจนภายในโมเลกุลของอาร์เอ็นเอหนึ่งๆ ได้หลายแบบ (ภาพที่ 2) นอกเหนือจากแบบของวัตสันและคริก เช่น แบบฮุกสทีน (Hoogsteen bbasepairing) หรือพันธะของเบสกับน้ำตาล (sugar edge) (ภาพที่ 3) นิวคลีโอไทด์ที่อยู่ห่างกันในโมเลกุลสามารถที่จะเกิดพันธะไฮโดรเจนกันแล้วม้วนขดเกิดโครงสร้างระดับทุติยภูมิ (secondary structure) และตติยภูมิ (tirtiary structure) ที่หลากหลายและแตกต่างไปจากโครงสร้างเกลียวคู่ของดีเอ็นเอได้
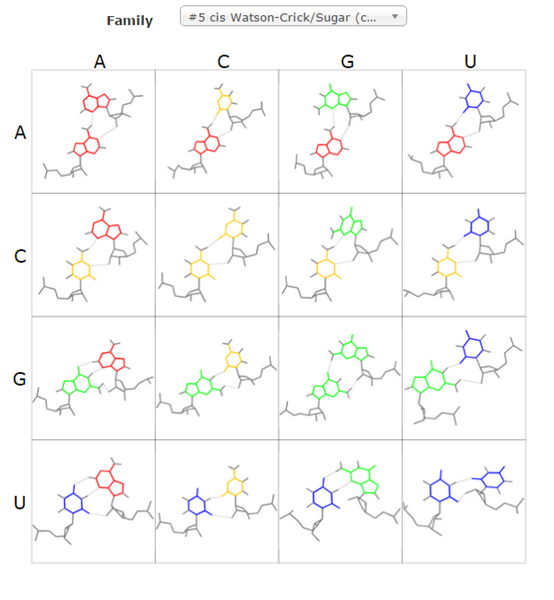
ภาพที่ 2 การเข้าคู่ของไนโตรจีนัสเบสที่นอกเหนือจากแบบของวัตสันและคริกในโมเลกุลของอาร์เอ็นเอ
นอกจากนี้ตำแหน่งของพันธะไกลโคซิดิก (cis หรือ trans) ที่เข้าทำพันธะไฮโดรเจนระหว่างไนโตรจีนัสเบสนั้นก็จะส่งผลต่อการเกิดโครงสร้างที่แตกต่างกันของอาร์เอ็นเอด้วย (ภาพที่ 3) ด้วยคุณสมบัติต่างๆ ดังกล่ามาแล้วจึงทำให้เกิดจับคู่ของไนโตรจีนัสเบสของอาร์เอ็นเอเกิดได้หลากหลายรูปแบบอย่างยิ่ง เช่น อะดีนีนกับอะดีนีน อะดีนีนกับไซโทซีน หรือ อะดีนีนกับกวานีน เป็นต้น ดังภาพที่ 2
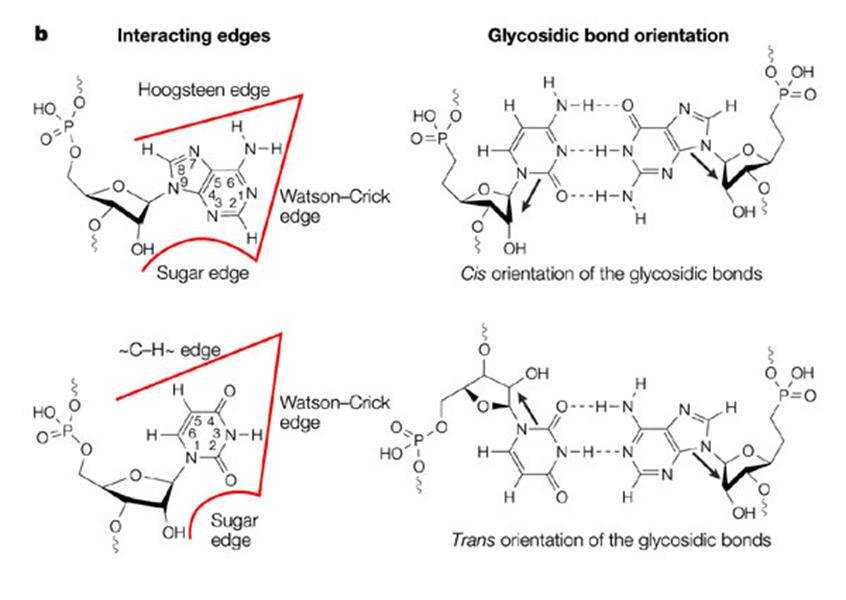
ภาพที่ 3 การเกิดพันธะไฮโดรเจนของไนโตรจีนัสเบสในโมเลกุลของอาร์เอ็นเอในรูปแบบต่างๆ
2. ฐานข้อมูลที่เกี่ยวข้องกับอาร์เอ็นเอ
การศึกษาค้นคว้าข้อมูลเกี่ยวกับอาร์เอ็นเอสามารถทำได้ผ่านฐานข้อมูลจำนวนมากในปัจจุบัน นอกจากฐานข้อมูล Rfam ซึ่งสามารถสืบค้นโดยใช้ข้อมูลลำดับนิวคลีโอไทด์ของอาร์เอ็นเอที่ไม่ทราบโครงสร้างหรือหน้าที่ได้ นอกจากนี้ยังมี
- ฐานข้อมูล NDB (Nucleic acid database) รวบรวมข้อมูลลำดับนิวคลีโอไทด์ โครงสร้างและหน้าที่ของอาร์เอ็นเอจากสิ่งมีชีวิตชนิดต่างๆ และโปรแกรมที่เกี่ยวข้องกับการวิเคราะห์อาร์เอ็นเอจำนวนมาก ได้แก่ RNAbow สำหรับแสดงภาพกราฟิกของโอกาสเกิดการจับคู่ระหว่างนิวคลีโอไทด์ในโมเลกุลของอาร์เอ็นเอ R3D Align เป็นโปรแกรมสำหรับเปรียบเทียบโครงสร้างสามมิติของอาร์เอ็นเอ
- ฐานข้อมูล RNAcentral (https://rnacentral.org/) ของ EMBL-EBI เป็นฐานข้อมูล integrative ที่รวบรวมฐานข้อมูลที่เกี่ยวข้องกับอาร์เอ็นเอกว่า 44 ฐานข้อมูลมารวมไว้ด้วยกัน เช่น 5SrRNAdb ฐานข้อมูลของ 5S ribosomal RNAs LncBase เป็นฐานข้อมูลเกี่ยวกับ long non-coding RNAs และ miRBase เป็นฐานข้อมูลเกี่ยวกับ miRNA (ภาพที่ 4) สามารถใช้ฐานข้อมูลนี้เพื่อค้นหาลำดับนิวคลีโอไทด์ของอาร์เอ็นเอที่สนใจหรือจากจีโนมของสิ่งมีชีวิตที่ต้องการได้
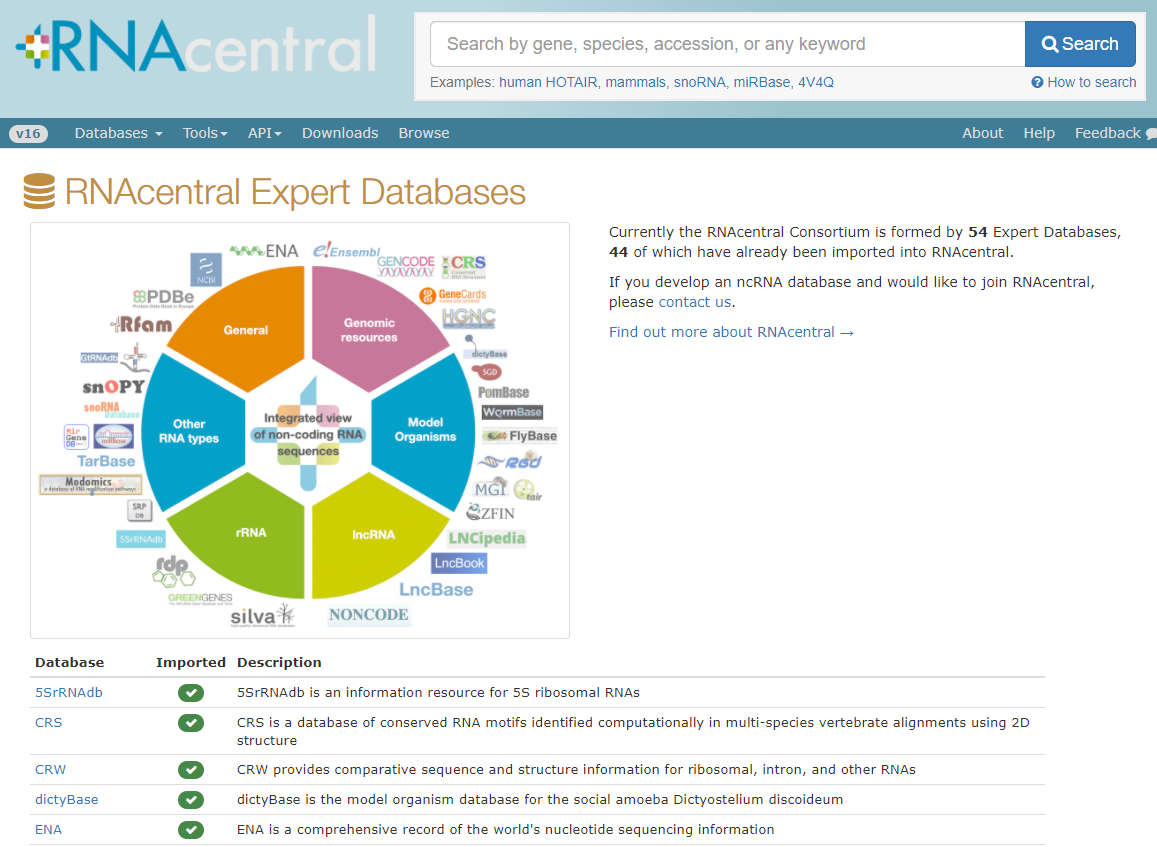
ภาพที่ 4 ฐานข้อมูล RNAcentral
3. การทำนายโครงสร้างขอองอาร์เอ็นเอ
เนื่องจากโมเลกุลของอาร์เอ็นเอมักเป็นสายเดี่ยวทำให้สามารถเกิดพันธะและโครงการที่หลากหลายได้ในสภาวะแวดล้อมของเซลล์ที่แตกต่างกัน การทำนายโครงสร้างของอาร์เอ็นเอจึงเป็นโจทย์ที่ท้าทายสำหรับนักชีวสารสนเทศ โครงสร้างของอาร์เอ็นเอแบ่ง 3 ระดับสำคัญคือ
- โครงสร้างระดับปฐมภูมิ (primary structure) คือ ลำดับของนิวคลีโอไทด์ที่ประกอบเป็นสายของอาร์เอ็นเอ
- โครงสร้างระดับทุติยภูมิ (secondary structure) คือ โครงสร้างที่เกิดจากการเข้าคู่ของนิวคลีโอไทด์ในตำแหน่งใกล้เคียงกัน
- โครงสร้างระดับตติยภูมิ (tertiary structure) คือ โครงสร้างสามมิติที่เกิดจากการเกิดพันธะระหว่างนิวคลีโอไทด์ที่มีตำแหน่งห่างไกลกันในโมเลกุล (distant interaction)
การทำความเข้าใจโครงสร้างของอาร์เอ็นเอซึ่งมีความซับซ้อนเริ่มจากการสร้างสัญลักษณ์แทนตำแหน่งของนิวคลีโอไทด์ในโมเลกุลของอาร์เอ็นเอที่มีการเกิดพันธะระหว่างกัน มีหลายวิธี (ภาพที่ 5) ได้แก่
- standard method โดยเขียนลำดับของตัวเลขแทนลำดับของนิวคลีโอไทด์ และแทนการจับคู่ของนิวคลีโอไทด์ด้วยการลากเส้นเชื่อม
- dot plot method ทำโดยเขียนลำดับนิวคลีโอไทด์ของสายอาร์เอ็นเอเส้นเดียวกันลงบนตารางทั้งในแนวคอลัมและแนวแถว ตำแหน่งของนิวคลีโอไทด์ที่เกิดการเข้าคู่กันจำระบายเปป็นแถบทึบหรือจุด
- Nussinov method ทำโดยแทนสายของอาร์เอ็นด้วยเส้นวงกลมและตำแหน่งของนิวคลีโอไทด์แทนด้วยจุด หากมีการเข้าคู่ของนิวคลีโอไทด์จะลากเส้นเชื่อจุดเข้าด้วยกัน
- dome method ใช้เส้นตรงแทนสายของอาร์เอ็นเอและจุดแทนนิวคลีโอไทด์ในตำแหน่งต่างๆ การเข้าคู่ของนิวคลีโอไทด์จะเขียนแทนด้วยเส้นโค้ง
- mountain method ใช้เส้นตรงแทนสายของอาร์เอ็นเอและจุดแทนนิวคลีโอไทด์ในตำแหน่งต่างๆ เช่นกัน แต่ตำแหน่งของนิวคลีโอไทด์ที่ไม่เกิดการเข้าคู่จะเขียนแทนด้วยเส้นแทยงและตำแหน่งที่มีการเข้าคู่จะเขียนแทนด้วยเส้นแนวนอน
- bracket method เป็นการเขียนสัญลักษณ์แทนโครงสร้างของอาร์เอ็นเอที่ง่ายที่สุดคือ เขียนแทนนิวคลีโอไทด์ที่ไม่มีการเข้าคู่ด้วยจุด . และแทนนิวคลีโอไทด์ที่เข้าคู่กันด้วยวงเล็บ ( ) ซึ่งแต่ละวงเล็บจะแทนนิวคลีโอไทด์แต่ละคู่
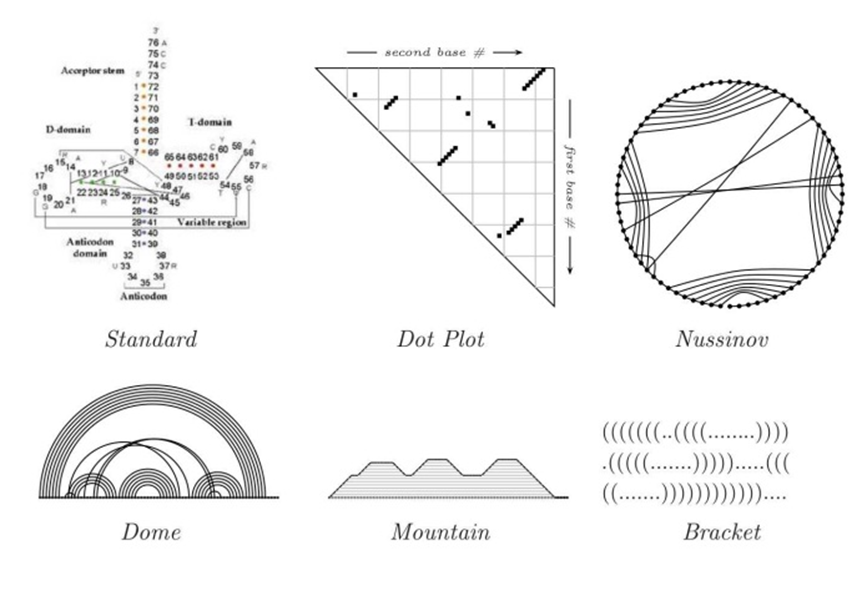
ภาพที่ 5 วิธีการเขียนสัญลักษณ์แทนการเข้าค่ของนิวคลีโอไทด์ในสายของอาร์เอ็นเอ
การเขียนสัญลักษณ์ตามวิธีดังกล่าวทำให้สามารถแสดงรูปแบบการเกิดโครงสร้างระดับทุติยภูมิของโมเลกุลอาร์เอ็นเอในเบื้องต้นได้ ดังภาพที่ 6
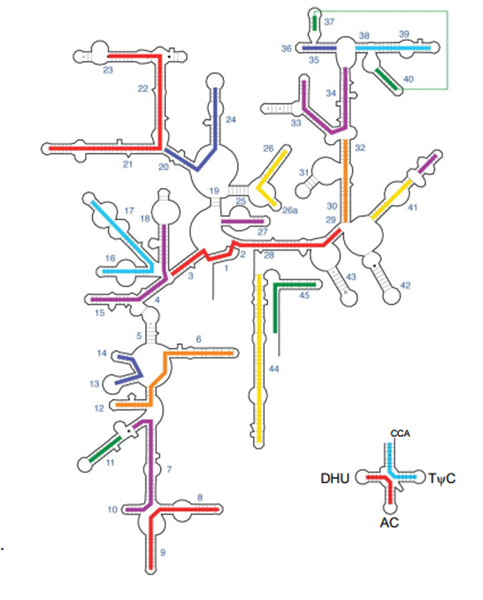
ภาพที่ 6 โครงสร้างระดับทุติยภูมิของ 16S rRNA
โครงสร้างระดับทุติยภูมิของอาร์เอ็นเอมีหลายรูปแบบ ได้แก่ ห่วงด้านปลาย (hairpin loop) ปมด้านเดียว (bulge) ห่วงแตกแขนง (multibranched loop) หรือบริเวณที่มีการเข้าคู่ของนิวคลีโอไทด์ติดต่อกันสองคู่ขึ้นไป (stem หรือ helix) ดังภาพที่ 7 การเกิดปฏิสัมพันธ์ของโครงสร้างเหล่านี้ที่มีตำแหน่งห่างกันในโมเลกุลจะทำให้เกิดโครงสร้างระดับตติยภูมิหรือโครงสร้างสามมิติของอาร์เอ็นเอขึ้น ได้แก่ ปมแบบซูโดน็อต (pseudoknot) ห่วงด้านปลายประกบกัน (kissing hairpins) หรือ ห่วงประกบกับปมด้านเดียว (hairpin-bulge) ดังภาพที่ 8
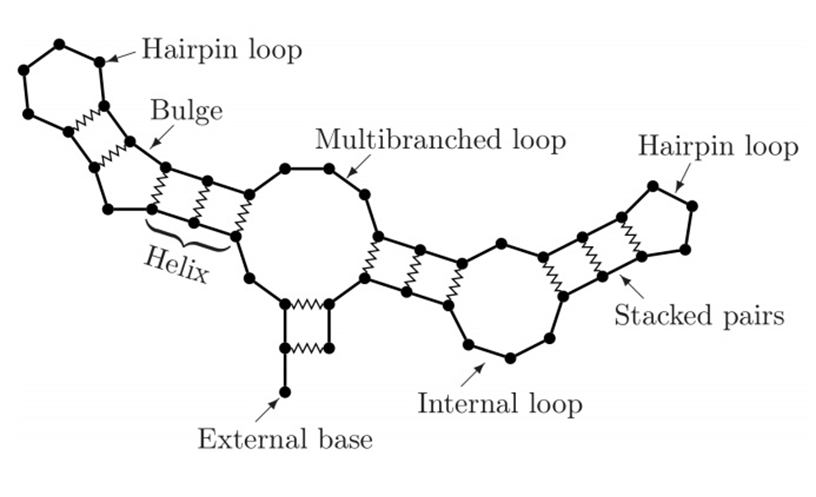
ภาพที่ 7 รูปแบบของโครงสร้างระดับทุติยภูมิในโมเลกุลของอาร์เอ็นเอ
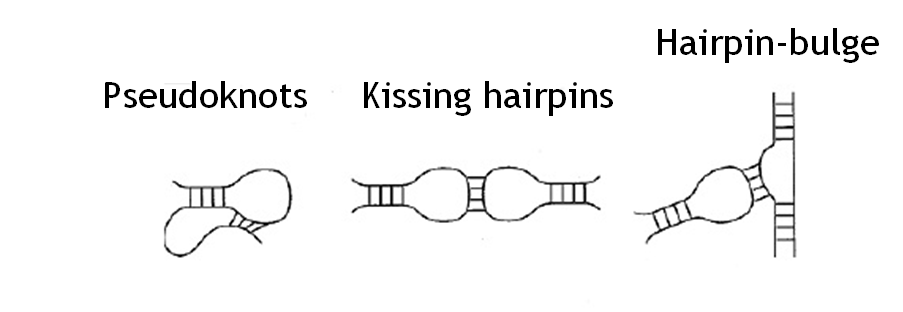
ภาพที่ 8 รูปแบบโครงสร้างระดับตติยภูมิของอาร์เอ็นเอ
การเกิดโครงสร้างระดับตติยภูมินี้อาจทำให้เกิดปฏิสัมพันธ์ระหว่างโมเลกุลของอาร์เอ็นเอหรือกับโมเลกุลอื่นๆ เช่น ดีเอ็นเอและโปรตีนได้ เกิดเป็นโครงสร้างระดับจตุรภูมิ (quaternary structure) ดังภาพที่ 9
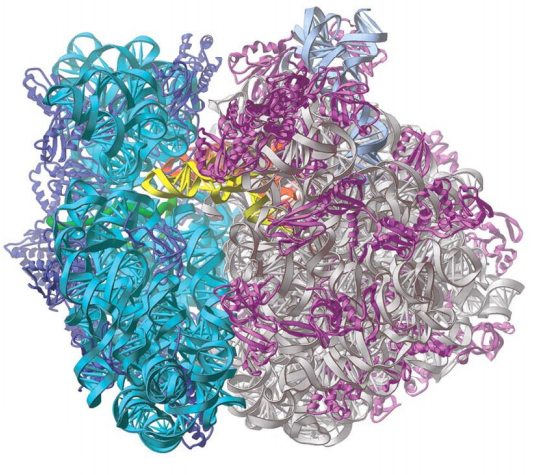
ภาพที่ 9 โครงสร้างระดับจตุรภูมิของ 70S ribosome ซึ่งประกอบด้วย rRNA 3 โมเลกุล กับโปรตีน 50 โมเลกุล
การทำนายโครงสร้างระดับทุติยภูมิและตติยภูมิของอาร์เอ็นเอมีหลายวิธี (Seetin and Mathews, 2012) ดังต่อไปนี้
- การเปรียบเทียบกับลำดับนิวคลีโอไทด์ที่ทรายโครงสร้างอยู่แล้ว (comparative sequence analysis) บนพื้นฐานว่าหากอาร์เอ็นที่สนใจกับอาร์เอ็นเอที่ทราบโครงสร้างอยู่แล้วนั้นเปลี่ยนแปลงมาจากบรรพบุรุษเดียวกัน (homologous sequence) โครงสร้างของอาร์เอ็นเอที่สนใจจะสามารถทำนายจากโครงสร้างของอาร์เอ็นเอต้นแบบได้ เช่น การทำนายโครงสร้างของไรโบโซมอลอาร์เอ็นเอ
- กรณีที่ไม่สามารถทำนายโครงสร้างของอาร์เอ็นเอจากโครงสร้างของอาร์เอ็นเอที่ร่วมบรรพบุุรุษมาด้วยกันได้ วิธีการทำนายโครงสร้างของอาร์เอ็นเอสามารถแบ่งออกเป็น
- การทำนายโครงสร้างของอาร์เอ็นเอโมเลกุลเดียว (single sequence structure prediction) มีหลายวิธีได้แก่ 1) การทำนายโครงสร้างของอาร์เอ็นเอที่มีค่าพลังงานอิสระต่ำสุด ณ อุณหภูมิและสารละลายที่กำหนด (free energy minimization) โดยทำนายโครงสร้างของอาร์เอ็นเอทุกแบบที่เป็นไปได้เพื่อเลือกโครงสร้างที่มีค่าพลังงานอิสระต่ำที่สุดให้เป็นโครงสร้างที่เป็นไปได้สูงสุดในสภาวะที่กำหนด 2) การทำนายโอกาสเกิดการเข้าคู่ของนิวคลีโอไทด์ด้วย partition function 3) การทำนาย maximum expected accuracy structure หรือโครงสร้างที่โอกาสถูกต้องมากที่สุดจากค่าโอกาสการเข้าคู่ของนิวคลีโอไทด์ และ 4) การทำนายโครงสร้างของอาร์เอ็นเอจากโครงสร้างระดับย่อย (suboptimal structure prediction) เช่น โปรแกรม MaxExpect และ RNAfold
- การทำนายโครงสร้างโดยใช้ข้อมูลจากการทดลองประกอบ (experimental mapping) เช่นการใช้เอนไซม์ไรโบนิวคลีเอส (ribonuclease) ที่มีตำแหน่งตัดจำเพาะบริเวณที่ไม่เกิดการเข้าคู่ของนิวคลีโอไทด์
- การทำนายโครงสร้างจากอาร์เอ็นเอหลายโมเลกุล (multile-sequence secondary structure prediction) โดยทำการเปรียบเทียบลำดับนิวคลีโอไทด์ของอาร์เอ็นเอหลายโมเลกุลก่อนแล้วทำนายโครงสร้างของอาร์เอ็นเอจากบริเวณอนุุรักษ์ (conserved region) เช่น โปรแกรม FOLDALIGN, Dynalign, LocARNA, Multilign, RNAalifold และ MC-FOLD
4. แบบฝึกการวิเคราะห์โครงสร้างและหน้าที่ของอาร์เอ็นเอ
ในกรณีที่มีโมเลกุลของอาร์เอ็นเอที่สนใจแต่ไม่ทราบหน้าที่ (accession number NR_144438.1) ในภาพที่ 10 อาจเริ่มต้นวิเคราะห์จากการนำลำดับนิวคลีโอไทด์ของอาร์เอ็นเอที่สนใจมาเปรียบเทียบกับฐานข้อมูลของอาร์เอ็นเอ เช่น ฐานข้อมูล Rfam ผลพบว่าอาร์เอ็นเอโมเลกุลนี้มีความคล้ายคลึงกับ long non-coding RNA ในหนูทั้งชนิด Rattus norvegicus และ Mus musculus โดยยังไม่มีข้อมูลโครงสร้างและไม่จัดอยู่แฟมิลี่ใดๆ ของฐานข้อมูล Rfam (ภาพที่ 11) อาร์เอ็นเอโมเลกุลนี้จึงอาจน่าสนใจในการวิเคราะห์และทำนายโครงสร้างต่อ
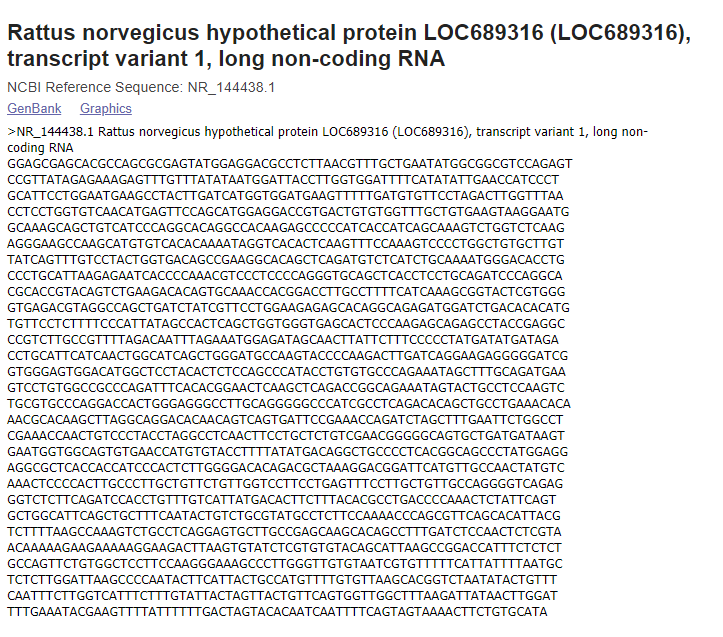
ภาพที่ 10 ตัวอย่างของอาร์เอ็นที่อาจไม่ทราบหน้าที่
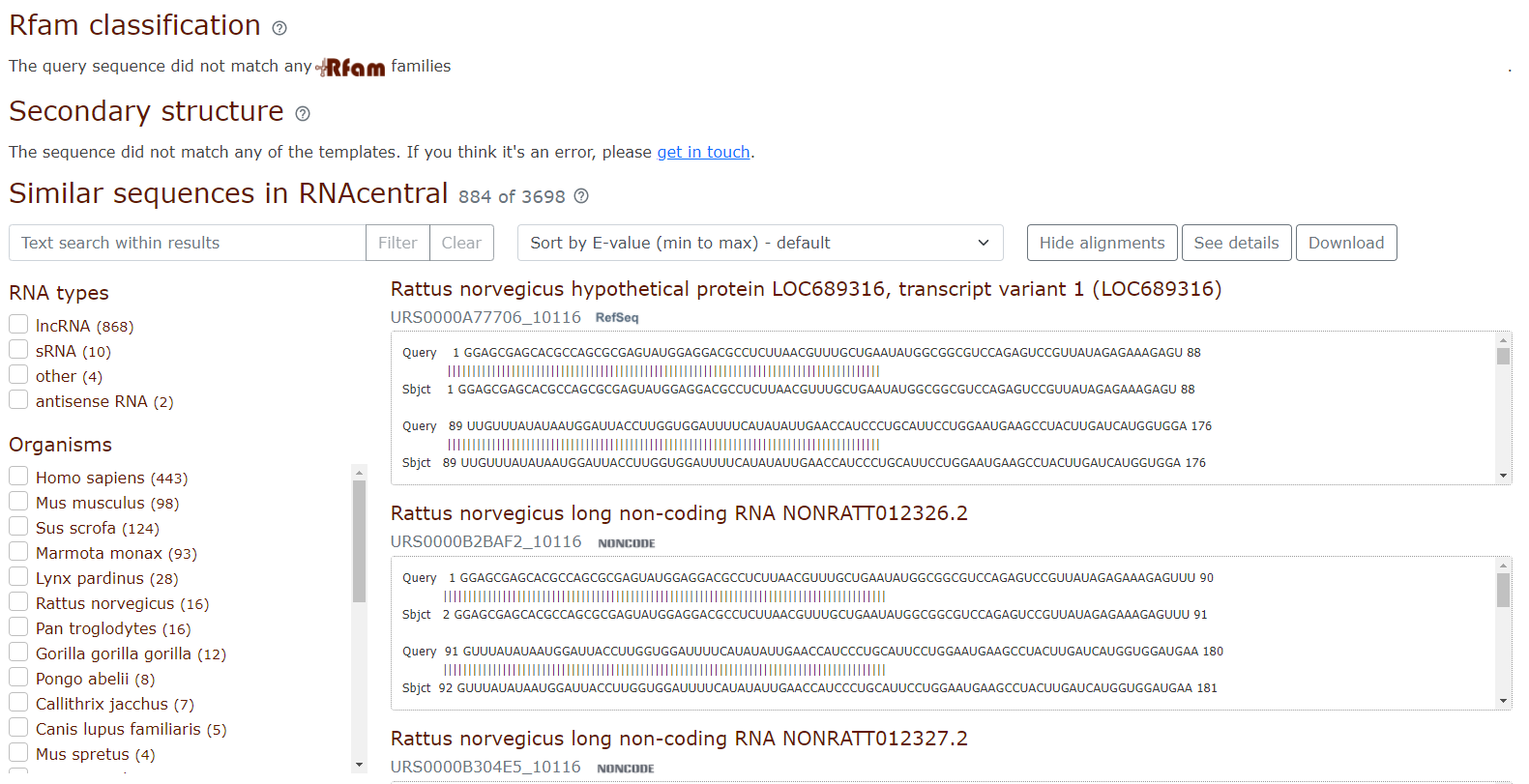
ภาพที่ 11 ผลการเปรียบเทียบอาร์เอ็นเอที่สนใจกับฐานข้อมูล Rfam
ในตัวอย่างนี้จะทำนายโครงสร้างทุติยภูมิของอาร์เอ็นเอโมเลกุลนี้ด้วยโปรแกรม RNAfold จากภาพที่ 12 โปรแกรมสามารถทำนายโครงสร้างของอาร์เอ็นเอตัวอย่างนี้ได้ด้วยวิธี minimum free energy (MFE) และ thermodynamic ensemble จะเห็นได้ว่าโครงสร้างจากวิธีการแรกจะมีค่าพลังงานอิสระต่ำกว่าจึงมีโอกาสเกิดโครงสร้างแบบนี้ได้มากกว่า (ภาพที่ 13) อย่างไรก็ตามอาร์เอ็นเอตัวอย่างนี้มีข้อมูลในฐานข้อมูลต่างๆ ให้เทียบเคียงน้อยมาก ทำให้ยังไม่สามารถทำนายหน้าที่ที่ชัดเจนได้ จำเป็นต้องมีการทดลองเพิ่มเติมเพื่อศึกษาหน้าที่หรือการทำงานของอาร์เอ็นเอโมเลกุลนี้ต่อไป การที่สามารถค้นหาอาร์เอ็นเอที่ไม่ทราบหน้าที่แล้ว (hypothetical RNA) ใช้การวิเคราะห์ทางชีวสารสนเทศช่วยเพิ่มเติมข้อมูลจะทำให้เกิดความเข้าใจในโครงสร้างและการทำงานของอาร์เอ็นเอมากยิ่งขึ้น การไม่พบข้อมูลหรือมีข้อมูลจำกัดจะเป็นแนวทางสู่การตั้งคำถามวิจัยและออกแบบการทดลองเพื่อพิสจน์ยืนยันต่อไปได้
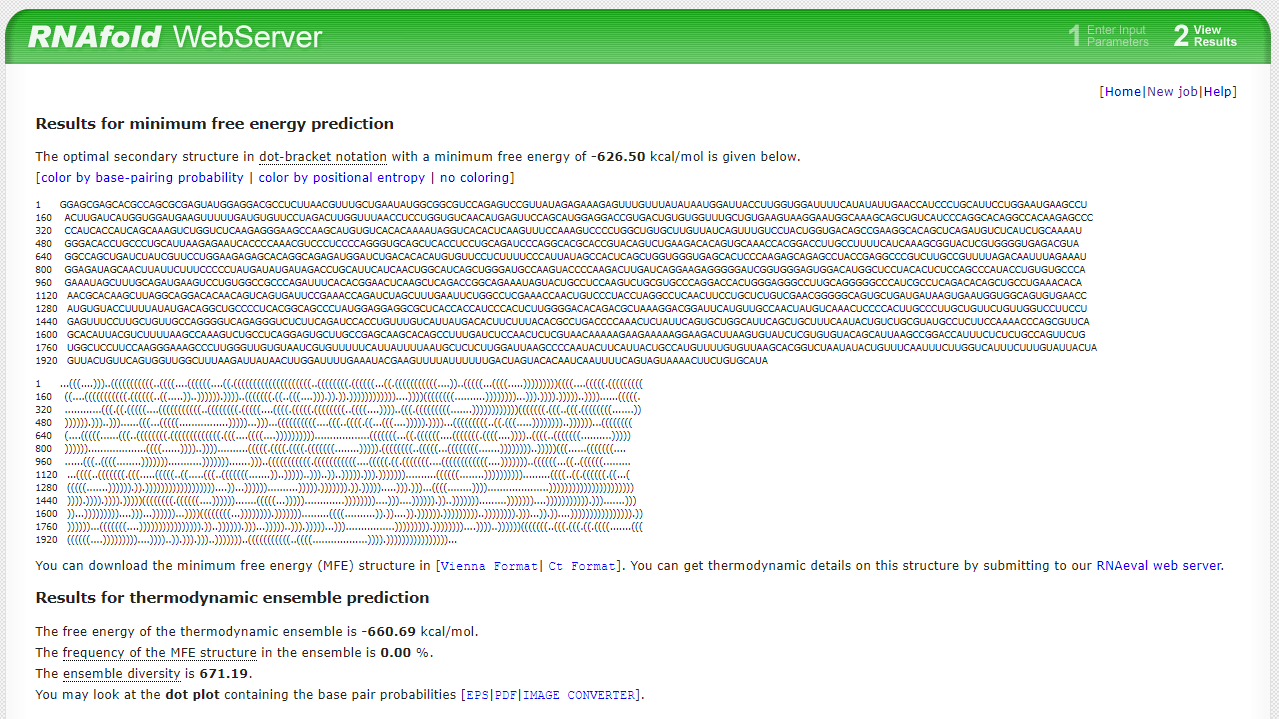
ภาพที่ 12 ผลการทำนายโครงสร้างของอาร์เอ็นเอที่สนใจด้วยโปรแกรม RNAfold
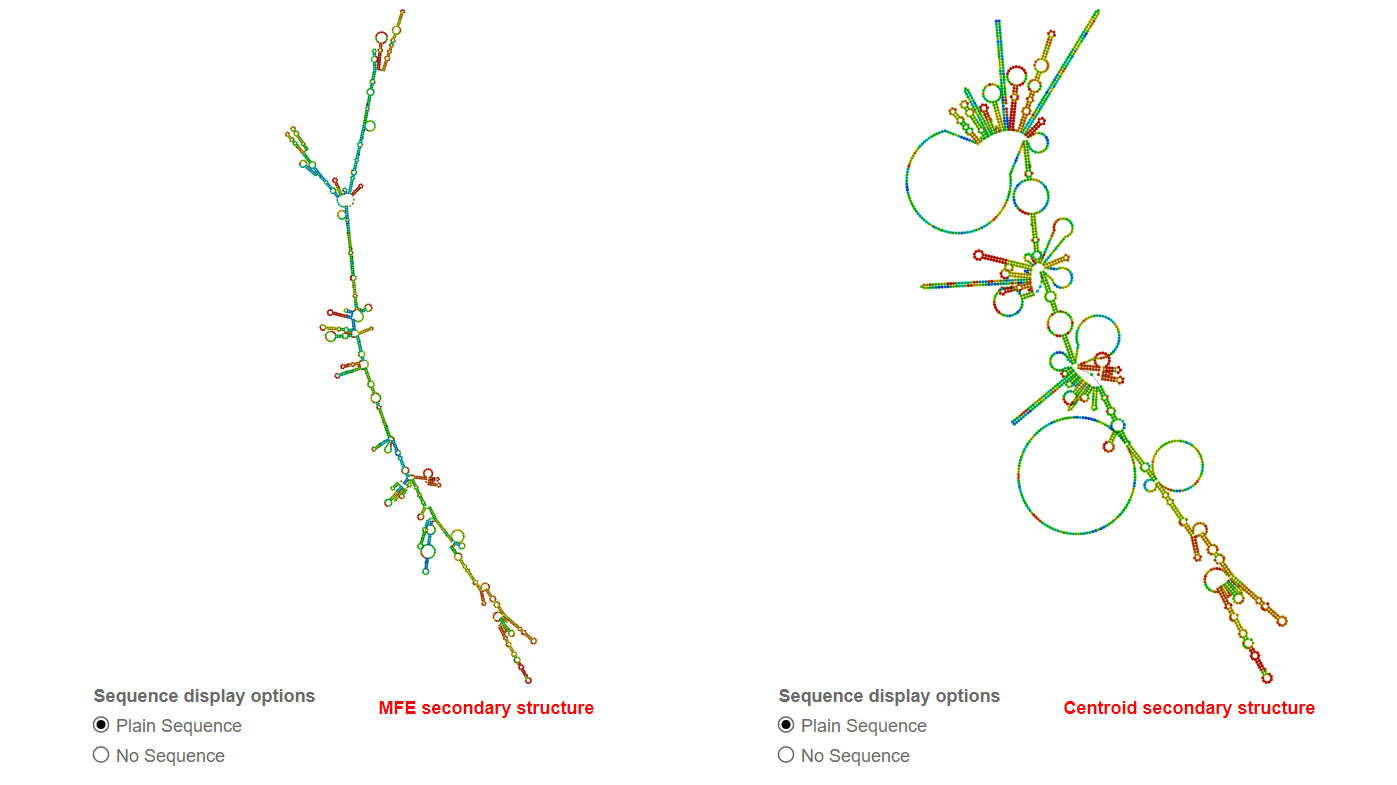
ภาพที่ 13 โครงสร้างระดับทุติยภูมิของอาร์เอ็นเอที่ทำนายได้ด้วยโปรแกรม RNAfold
ตัวอย่างถัดมามี snoRNA ที่สนใจจาก Saccharomyces cerevisiae (NR_132193.1) แล้วต้องการทราบข้อมูลเกี่ยวกับหน้าที่และโครงสร้าง เมื่อนำไปค้นกับฐานข้อมูล Rfam พบว่าเป็นอาร์เอ็นเอในแฟมิลี่ small nucleolar RNA SNORD14 (RF00016) มีหน้าที่สำคัญในขั้นตอนเริ่มต้นของการตัด rRNA ตั้งต้นเพื่อให้พร้อมทำงาน (ภาพที่ 14)
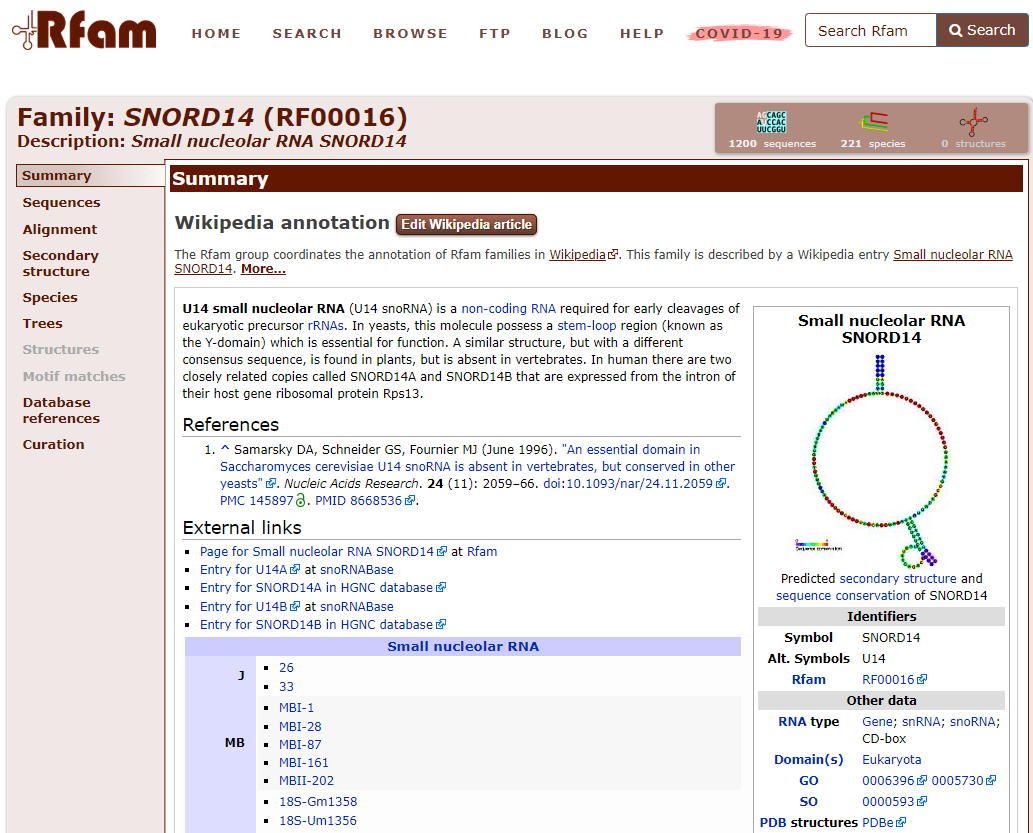
ภาพที่ 14 ผลการค้นฐานข้อมูล Rfam ด้วยตัวอย่างของ snoRNA
นอกจากนี้ในฐานข้อมูล Rfam ยังมีโปรแกรม R2DT สำหรับทำนายโครงสร้างระดับทติยภูมิโดยใช้โครงสร้างของอาร์เอ็นเอที่อาจมีบรรพบุรุษร่วมหรือคล้ายกับอาร์เอ็นเอที่สนใจเป็นต้นแบบด้วย จากโครงสร้างในภาพที่ 15 มีการแสดงสีของนิวคลีโอไทด์ที่แตกต่างกัน โดยสีดำ หมายถึงตำแหน่งและของนิวคลีโอไทด์นั้นเหมือนต้นแบบ สีเขียว คือ นิวคลีโอไทด์ในตำแหน่งนั้นแตกต่างจากต้นแบบ สีแดง คือลำดับนิวคลีโอไทด์เพิ่มขึ้นมาจากต้นแบบ และสีน้ำเงิน คือ ตำแหน่งของนิวคลีโอไทด์หรือโครงสร้างมีการเปลี่ยน หากเปรียบเทียบกับโครงสร้างของอาร์เอ็นเอต้นแบบในภาพที่ 14 จะเห็นว่ามีโครงสร้างที่ไม่เหมือนกัน โครงสร้างในภาพที่ 15 ประกอบด้วย 1 hairpin loop มี buldge ขนาดใหญ่ตรงกลางและมี stem ที่ปลายอีกด้านหนึ่ง ซึ่งข้อมูลโครงสร้างนี้อาจนำไปใช้ประกอบการอธิบายผลจากการทดลองหรือวิจารณ์ผลการทดลองเพื่อเปรียบเทียบกับข้อมูลที่มีอย่ก่อนหน้าแล้วได้
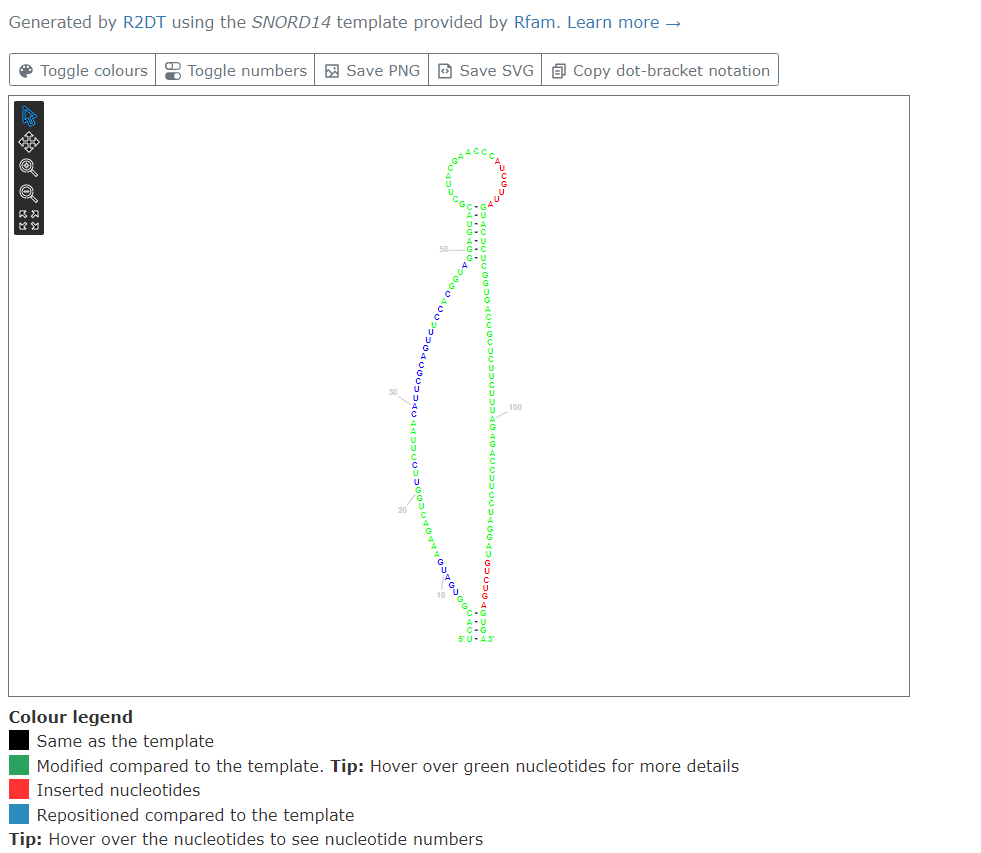
ภาพที่ 15 ผลการทำนายโครงสร้างของ snoRNA ที่สนใจ
จากข้อมูลโครงสร้างและหน้าที่ที่ทราบเบื้องต้นในสิ่งมีชีวิตชนิดนี้แล้วนั้น นักวิจัยอาจตั้งคำถามต่อมาถึงความแตกต่างของโครงสร้างและหน้าที่ในสิ่งมีชีวิตต่างชนิดกัน สามารถทำการวิเคราะห์เปรียบเทียบลำดับนิวคลีโอไทด์และโครงสร้างต่อไปได้ หรือถ้าต้องการทำนายโครงสร้างสามมิติของอาร์เอ็นเอโมเลกุลนี้สามารถทำได้โดยใช้โปรแกรม iFoldRNA v2 (ภาพที่ 16)
ภาพที่ 16 ตัวอย่างโปรแกรมสำหรับการทำนายโครงสร้างสามมิติของอาร์เอ็นเอ
5. การออกแบบโมเลกุุลอาร์เอ็นเอผ่านเกม Eterna
เกมนี้ออกแบบมาเพื่อให้คนทั่วโลกไม่จำกัดอายุสามารถเรียนรู้เกี่ยวกับโครงสร้างของอาร์เอ็นเอและร่วมทดลองออกแบบโครงสร้างของอาร์เอ็นเอด้วยคอมพิวเตอร์ส่วนบุคคลได้ โดยผู้เล่นจะสร้างบัญชีผู้ใช้งาน จะมีด่านหรือ puzzle ให้ผู้เล่นได้ทดลองออกแบบโมเลกุลของอาร์เอ็นเอให้ได้โครงสร้างหรือมีระดับพลังงานอิสระที่ต่ำพอจะเกิดโครงสร้างนี้ในสภาวะที่กำหนดได้ (ภาพที่ 17)
ภาพที่ 17 หน้าต่างแรกของเกม Eterna
ในระหว่างการเล่นเกมนี้จะมีคำอธิบายและความรูู้เกี่ยวกับอาร์เอ็นเอให้ผู้เล่นได้เข้าใจข้อความรู้ระหว่างการเล่นเกม บางโจทย์จะเป็นโมเลกุลของอาร์เอ็นเอจากสิ่งมีชีวิตจริงเช่น อาร์เอ็นเอที่เกี่ยวข้องกับการควบคุมโรคต่างๆ หรืออาร์เอ็นเอที่ต้องการออกแบบเพื่อใช้ในการยับยั้งการทำงานของไวรัสที่ก่อโรค นอกจากนี้จะมีการสะสมคะแนนของผู้เล่นและจัดลำดับ (ภาพที่ 18 และ 19) รวมทั้งหากโครงสร้างอาร์เอ็นเอของผู้เล่นคนใดมีความน่าสนใจ ทีมวิจัยผู้พัฒนาเกมนี้อาจนำไปสังเคราะห์หรือออกแบบการทดลองเพื่อทดสอบโครงสร้างนั้นต่อไปได้ด้วย
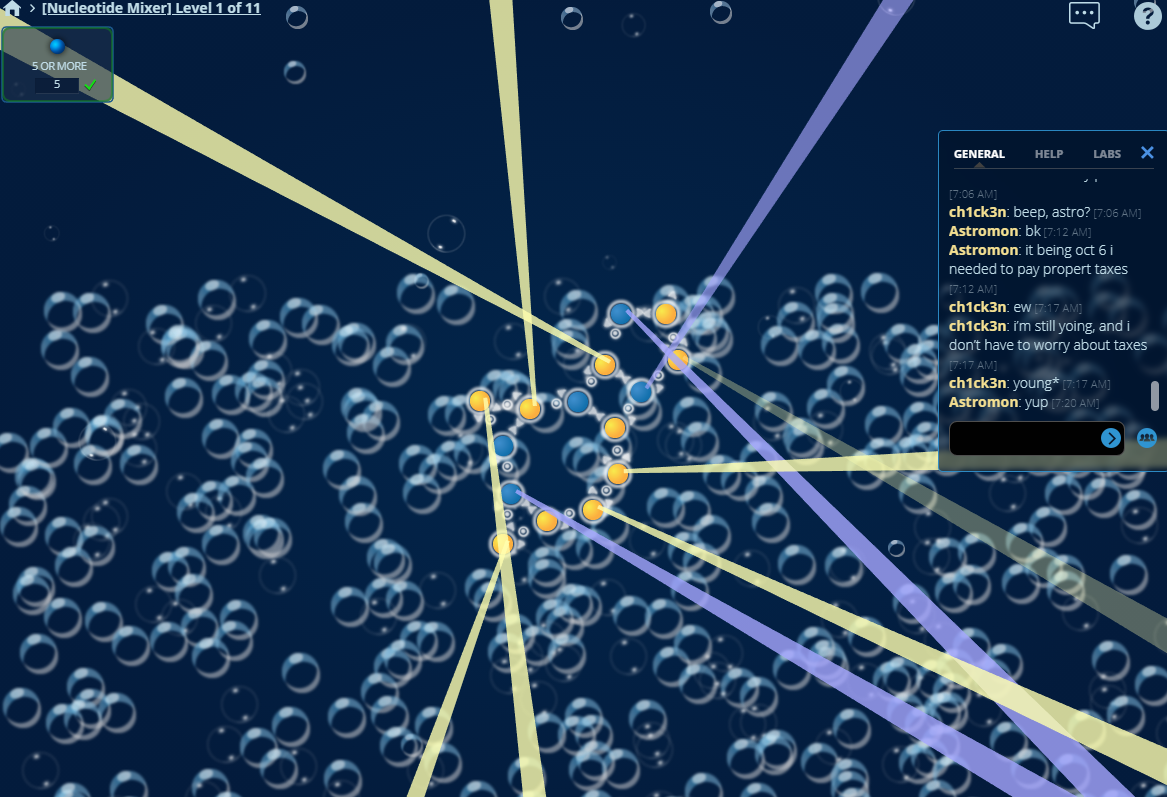
ภาพที่ 18 ตัวอย่างของการออกแบบโครงสร้างของอาร์เอ็นเอผ่านเกม Eterna

ภาพที่ 19 ภารกิจต่างๆ ในเกม Eterna ที่ผู้เล่นสามารถเลือกได้
จากตัวอย่างข้างต้นแสดงให้เห็นถึงความก้าวหน้าและความสำคัญของชีวสารสนเทศต่อการศึกษาและวิจัยหน้าที่และโครงสร้างของอาร์เอ็นเอ นำสู่คำถามวิจัยใหม่หรือแนวทางในการพัฒนาเทคโนโลยีเพื่อใช้จริงในชีวิตประจำวัน
