ปัจจุบันเทคโนโลยี next generation sequencing (NGS) มีบทบาทต่อการศึกษาวิจัยทางชีววิทยาเป็นอย่างมาก เทคโนโลยี high throughput นี้สามารถวิเคราะห์ลำดับนิวคลีโอไทด์ของดีเอ็นเอหรืออาร์เอ็นเอได้หลายล้านหรือพันล้านเส้นพร้อมกัน (parallel sequencing) เทคโนโลยี NGS ช่วยให้นักวิจัยวิเคราะห์ข้อมูลจีโนมของสิ่งมีชีวิตได้รวดเร็วขึ้น จีโนมประกอบขึ้นจากสารพันธุกรรมทั้งหมดของสิ่งมีชีวิต เรียกว่า whole genome การเลือกศึกษาเฉพาะส่วนของจีโนมที่เกี่ยวข้องกับยีนหรือ exon นั้นเรียกว่า exome ซึ่งขนาดของข้อมูลเล็กกว่าจีโนมและราคาประหยัดกว่า
การเตรียมตัวอย่างดีเอ็นเอเพื่อการศึกษาจีโนมหรือเอกโซมด้วยวิธี short read sequencing นั้นเริ่มจาก การเตรียม genomic DNA library โดยทำให้สายดีเอ็นเอขาดเป็นเเส้นสั้นประมาณ 200-600 เบส ดำเนินการ repair ends และติด adaptors หรือ barcode เพื่อให้ผสม DNA library จากตัวอย่างที่ต่างกันได้ (multiplexing) หากต้องการศึกษาเอกโซม จะต้องทำการ capture fragments ที่มีส่วนของเอกซอนอยู่ก่อน แล้วจึงดำเนินการวิเคราะห์ลำดับนิวคลีโอไทด์ของ genomic DNA library ที่เตรียมได้ เช่น เทคโนโลยีของ Illumina ที่วิเคราะห์ลำดับนิวคลีโอไทด์ด้วยการสังเคราะห์สายใหม่จากต้นแบบของ library ที่ใช้ (sequencing by synthesis) โดยเครื่อง sequencer ชิ้นส่วนของดีเอ็นเอแต่ละเส้นใน library จะผ่านปฏิกิริยาการสังเคราะห์สายใหม่ไปพร้อมๆ กัน เมื่อเติมนิวคลีโอไทด์ต่างชนิดเข้าไปในสายใหม่จะเกิดสีที่ต่างกัน ซึ่งโปรแกรมจะบันทึกภาพการเปลี่ยนแปลงสีของแต่ละปฏิกิริยาเอาไว้เพื่อแปลผลออกมาเป็นลำดับนิวคลีโอไทด์แต่ละเส้นใน library ที่ใช้ (image analysis)
ข้อมูลที่ได้จะเป็นสายดีเอ็นเอขนาดสั้น (sequence reads) จำนวนมากในรูปแบบไฟล์ fastq จะนำมาประกอบ (assembly) กลับมาเป็นสายดีเอ็นเอเส้นยาวด้วยวิธีการที่ใช้จีโนมอ้างอิง (reference-based assembly หรือ read mapping) เมื่อมี coverage หรือ sequencing depth ซึ่งหมายถึงจำนวนซ้ำของ reads ที่ตรงกับลำดับนิวคลีโอไทด์ตำแหน่งนั้นๆ มากพอ สามารถวิเคราะห์เปรียบเทียบความแตกต่างระหว่างจีโนมที่สนใจกับจีโนมอ้างอิง ระบุความผันแปรที่เกิดขึ้นในรูปแบบต่างๆ ได้แก่ single nucleotide polymorphisms (SNPs) insertion และ deletion ขนาดเล็ก (indels) structural variations (SV) copy number variations (CVs) และ repeats ขั้นตอนการวิเคราะห์สรุปดังภาพที่ 1
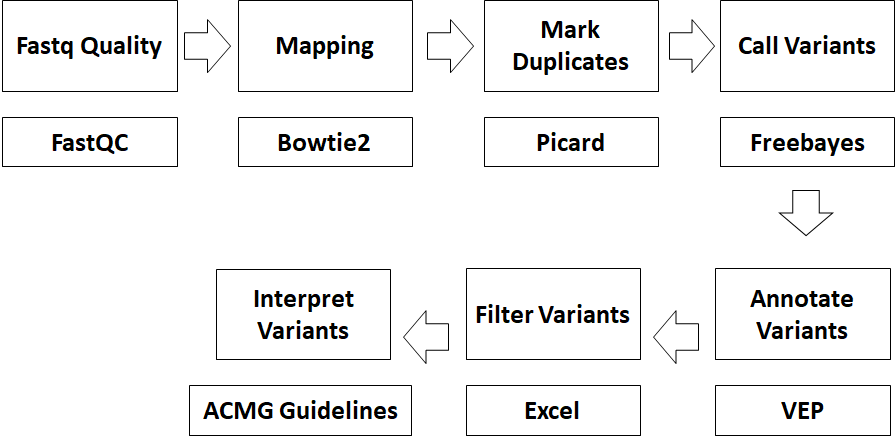
การวิเคราะห์ข้อมูล NGS สามารถเริ่มต้นเรียนรู้การวิเคราะห์ได้โดยใช้โปรแกรม Galaxy ซึ่งเป็น web-based platform มีโปรแกรมสำหรับวิเคราะห์ให้ใช้งานได้จำนวนมากโดยไม่ต้องทำการติดตั้งบนคอมพิวเตอร์ การวิเคราะห์ข้อมูล NGS ในครั้งนี้จะใช้ข้อมูล exome ในบริเวณของยีน BRCA2 จากผู้ป่วยรายหนึ่งเป็นตัวอย่างในการศึกษา
การตรวจสอบคุณภาพของไฟล์ fastq
เริ่มต้นโดยลงทะเบียนเพื่อใช้งาน Galaxy ที่ https://usegalaxy.org/ เมื่อเข้าสู่โปรแกรมแล้วจะพบแถบด้านซ้ายซึ่งแสดงรายชื่อของโปรแกรมต่าง ๆ จัดกลุ่มตามประเภทของการวิเคราะห์ พื้นที่ตรงกลางจะเป็นบริเวณที่ใช้ปรับแต่งค่าพารามิเตอร์สำหรับใช้งานแต่ละโปรแกรมที่เลือกมา ส่วนแถบด้านขวามือจะแสดงประวัติและสถานะการทำงานของแต่ละโปรแกรม ดังภาพที่ 2
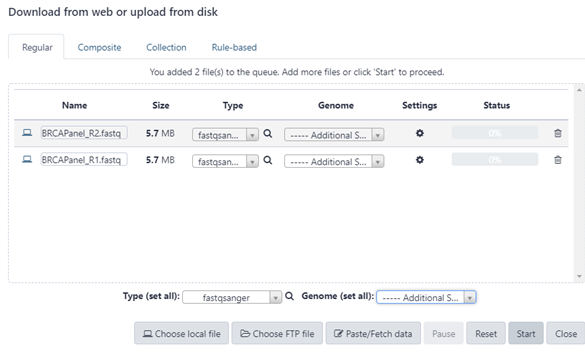
ไฟล์ fastq ตัวอย่างที่ใช้ในการวิเคราะห์สามารถโหลดได้จาก Google drive ของบทเรียนนี้ ไฟล์ fastq เป็นไฟล์ที่เก็บข้อมูลลำดับนิวคลีโอไทด์ของ sequence reads และค่าคะแนนคุณภาพของนิวคลีโอไทด์ในแต่ละตำแหน่ง (quality scores) ข้อมูลตัวอย่างประกอบด้วยไฟล์ _F.fastq และ _R.fastq นำไฟล์ทั้งสองเข้าสู่ Galaxy โดยเลือก Get Data > Upload File เลือก Choose local file เพื่อ upload ไฟล์ตัวอย่าง เลือก Type (set all) เป็น fastasanger จากนั้นคลิก start (ภาพที่ 3)
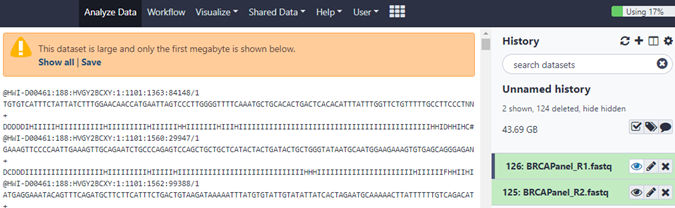
ในบรรทัดแรกของไฟล์ fastq จะเป็น identifier ซึ่งแสดงรายละเอียดที่จำเพาะกับ sequence read แต่ละเส้น บรรทัดที่สองจะเป็นลำดับนิวคลีโอไทด์ของ sequence read คั่นด้วยบรรทัดที่มีเครื่องหมายบวก (+) แล้วบรรทัดสุดท้ายจะเป็นค่า quality score (Q score) ที่บันทึกด้วยตัวอักษร ASCII ซึ่งตัวอักษรเหล่านี้จะมีค่าตั้งแต่ 33 ค่า Q score บอกถึงความเป็นไปได้ที่การอ่านลำดับนิวคลีโอไทด์ ณ ตำแหน่งใดตำแหน่งหนึ่งในสายจะผิดพลาด (base-calling probability) (ภาพที่ 4)
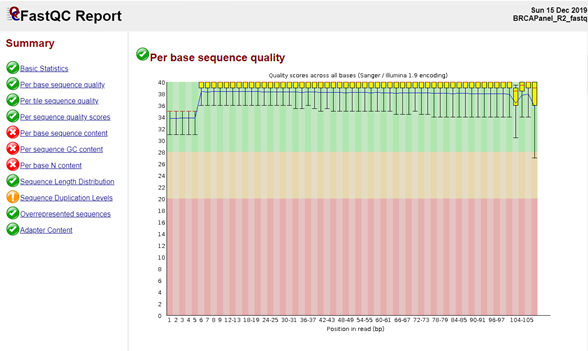
การตรวจสอบคุณภาพของ sequence reads ใช้โปรแกรม FastQC ในหมวด GENOME FILE MANIPULATION เลือก FASTQ Quality Control > FastQC เลือกไฟล์ fastqc ที่นำเข้ามาทีละหนึ่งไฟล์ แล้วคลิก execute สถานะการทำงานของโปรแกรมจะแสดงอยู่ทางด้านขวามือ ตรวจสอบผลได้โดยคลิกที่สัญลักษณ์รูปดวงตา จะเห็นว่าค่าคุณภาพในแต่ละตำแหน่งของ sequence reads ในไฟล์ทั้งสอง (per base sequence quality) มีค่าอยู่ในพื้นที่สีเขียวที่ค่า Q score ตั้งแต่ 28 ขึ้นไปแสดงว่าข้อมูลของ sequence reads ไฟล์นี้ในเบื้องต้นมีคุณภาพที่ดี (ภาพที่ 5) สามารถนำไปวิเคราะห์ในขั้นต่อไปได้ นอกจากนี้ยังสามารถพิจารณาค่าคุณภาพต่อสายของดีเอ็นเอ (per sequence quality scores) องค์ประกอบของนิวคลีโอไทด์ (per base sequence content) และอื่นๆ ได้ เพื่อเลือกวิธีแก้ไขหรือปรับข้อมูลเช่น ตัดส่วนที่คุณภาพไม่ดีออก (trimming) หรือตัดส่วนของ adapter ที่ติดมาด้วย ให้มั่นใจในคุณภาพก่อนวิเคราะห์ขั้นต่อไป
การเทียบ sequence reads กับจีโนมอ้างอิง (sequence read mapping)
เมื่อตรวจสอบคุณภาพของไฟล์ fastq แล้ว จะทำการเปรียบเทียบข้อมูล sequence reads ของตัวอย่างผู้ป่วยกับข้อมูลจีโนมอ้างอิงของมนุษย์ (human reference genome) เรียงว่า sequence read mapping หรือ alignment เพื่อระบุว่า sequence read แต่ละเส้นนั้นอยู่ตรงส่วนใดของจีโนม โดยมีหลายโปรแกรมพัฒนาข้นเพื่อการวิเคราะห์ขั้นนี้ได้แก่ Bowtie และ BWA
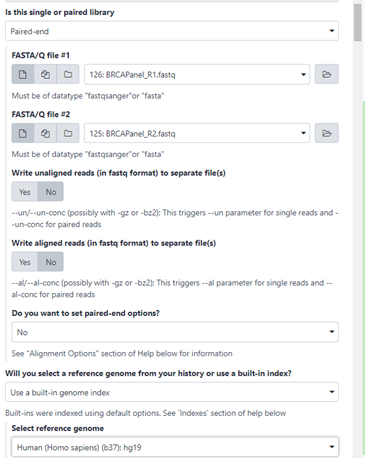
เริ่มต้นจากไปหมวด GENOMICS ANALYSIS > mapping > Bowtie2 เลือก Paired-end สำหรับ library แล้วเลือกไฟล์ตัวอย่าง (_R1 และ _R2) ทั้งสองไฟล์ และเลือก Human (Homo sapiens) hg19 เป็น reference genome จากนั้นคลิก execute (ภาพที่ 6)
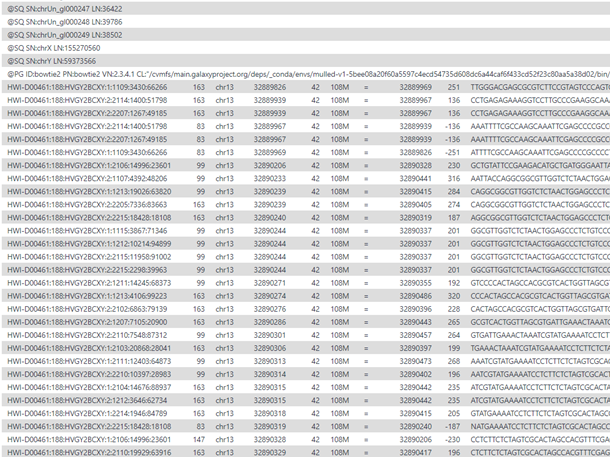
ผลที่ได้จาก read mapping จะบันทึกอยู่ในไฟล์ .sam (sequence alignment map) หรือ .bam ที่มาจากการย่อไฟล์ sam ให้มีขนาดเล็กลง โดยส่วนต้นของไฟล์จะแสดง header ประกอบด้วยชื่อของ sequence (SN) และความยาว (LN) ส่วนต่อมาในไฟล์นี้จะแสดงรายละเอียดของ alignment โดยประกอบด้วยข้อมูล 11 คอลัม คือ QNAME (sequence ID) FLAG (ค่าคุณภาพของ sequence หรือ sequence quality) RNAME (โครโมโซม) POS (ตำแหน่งเริ่มต้นหรือ start position) MAPQ (ค่าคุณภาพของการเปรียบเทียบหรือ mapping quality) CIGAR (อธิบายลักษณะของตำแหน่งนั้นว่าเหมือนหรือต่างจากอ้างอิงอย่างไร) RNEXT (Ref name of mate/next read) PNEXT (position of mate/next read) TLEN (observed template length) SEQ (ลำดับนิวคลีโอไทด์) และ QUAL (base qualities) (ภาพที่ 7)
ค่า FLAG ในคอลัมที่สองบอกถึงคุณภาพของ sequence read ว่าสามารถ map กับจีโนมอ้างอิงในรูปแบบใด ได้แก่
- 69 (The read is paired, is the first read in the pair, and is unmapped.)
- 77 (The read is paired, is the first read in the pair, both are unmapped.)
- 83 (The read is paired, mapped in a proper pair, is the first read in the pair, and it is mapped to the reverse strand.)
- 99 (The read is paired, mapped in a proper pair, is the first read in the pair, and its mate is mapped to the reverse strand.)
- 133 (The read is paired, is the second read in the pair, and it is unmapped.)
- 137 (The read is paired, is the second read in the pair, and it is mapped while its mate is not.)
- 141 (The read is paired, is the second read in the pair, but both are unmapped)
- 147 (The read is paired, mapped in a proper pair, is the second read in the pair, and mapped to the reverse strand.) และ
- 163 (The read is paired, mapped in a proper pair, is the second read in the pair, and its mate is mapped to the reverse strand.)
ค่า CIGAR (compact idiosyncratic gapped alignment report) ใช้สำหรับบอกรูปแบบการ map ของ read บนจีโนมอ้างอิง โดย
- M คือ เหมือนกัน (alignment match)
- I คือ มีชิ้นส่วนเกินมา (insertion)
- D คือ มีชิ้นส่วนหายไป (deletion)
- N คือ ข้ามหรือไม่ตรงกัน (skipped)
- S คือ มีบางส่วนของ read นี้ที่ map ไม่ต่อเนื่องกับจีโนมอ้างอิง (soft-clipped)
- H คือ มีบางส่วนของ read นี้ที่ map ไม่ต่อเนื่องกับจีโนมอ้างอิงและถูกตัดออกไป (hard-clipped)
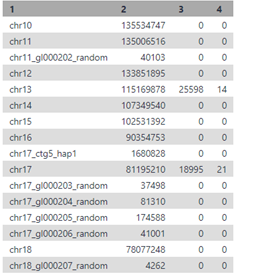
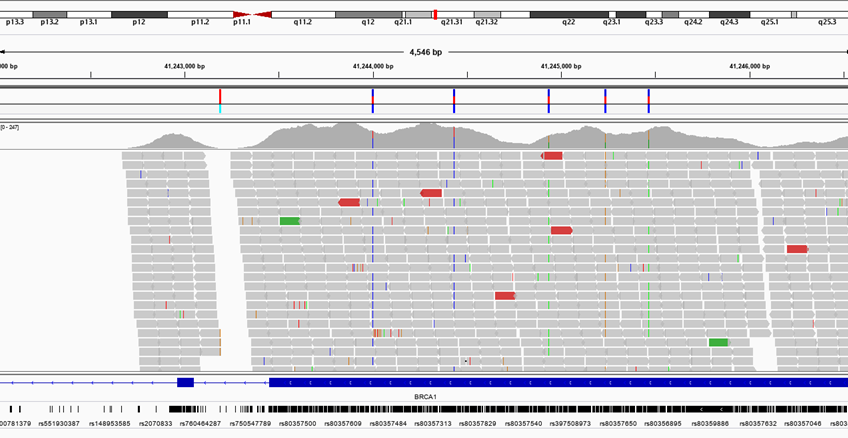
จากไฟล์ .bam ที่ได้ ขั้นต่อมาทำการสรุปผลของการทำ mapping โดยไปที่ SAM/BAM เลือก Samtools flagstat เพื่อสรุปผลการเปรียบเทียบออกมาเป็นตาราง ผลที่ได้จะบอกให้ทราบว่ามี 44,708 reads ที่ map กับจีโนมอ้างอิงได้สำเร็จ รวมถึง duplicates ซึ่งเกิดจากขั้นการทำ PCR ระหว่างเตรียม library ทำให้บาง read นั้นมีจำนวนมากเกินปกติ รวมทั้ง reads ที่เข้าคู่กันได้ (paired) หรือไม่ได้ (unpaired) จากนั้นเลือกโปรแกรม Samtools idxstats เพื่อสรุปว่า reads จากตัวอย่างสามารถ map ได้กับโครโมโซมใดบ้าง ผลปรากฎว่าส่วนใหญ่พบบนโครโมโซมคู่ที่ 13 และ 17 (ภาพที่ 8)
การตรวจสอบ duplicates สามารถทำได้ด้วยโปรแกรม Picard โดยไปที่ Picard เลือก MarkDuplicates ใช้ไฟล์ .bam เป็น Input จากนั้นสรุปผลด้วย Samtools flagstat อีกครั้ง โดยใช้ไฟล์ .bam ที่ได้มาจากโปรแกรม Picard ผลที่ได้จะพบว่ามี duplicates จำนวน 7,568 reads เพื่อดูผลในไฟล์ .bam นี้ด้วยโปรแกรมแสดงผล ให้คลิกที่เครื่องหมายแถบบันทึกทางด้านขวาเพื่อดาวน์โหลดไฟล์ .bam และ .bai ซี่งเป็นไฟล์ index ของไฟล์ .bam
การแสดงผลรายละเอียดในไฟล์ .bam ทำได้ด้วยโปรแกรม IGV โดยโปรแกรม IGV จะมีจีโนมอ้างอิงของมนุษย์ hg19 ติดตั้งอยู่เรียบร้อยแล้ว รวมทั้งสามารถโหลดข้อมูลเพิ่มเติมจากฐานข้อมูล เช่น dbSNPs มาประกอบได้ โดยเลือก FIle > Load from server เลือก Annotations > Variation and Repeats หรือฐานข้อมูลอื่นๆ ได้ โปรแกรมนี้ช่วยให้สามารถดูผลจากแต่ละโครโมโซม (เลือกจาก drop-down menu ด้านบน) ตลอดจนสามารถขยายภาพเพื่อดูรายละเอียดในระดับนิวคลีโอไทด์ได้ (กดสัญลักษณ์เครื่องหมาย + หรือ -) นอกจากนี้สามารถใช้กล่องค้นหาเพื่อแสดงบริเวณของยีนที่สนใจได้ เช่น BRCA1
เมื่อโปรแกรมพร้อม ทำการนำไฟล์ .bam เข้าสู่โปรแกรม โดยเลือก File > Load from file เมื่อขยายภาพจะเห็นข้อมูลของ mapped reads เมื่อเทียบกับตำแหน่งของจีโนมอ้างอิง (ภาพที่ 9) และสำรวจดูความผันแปรทางพันธุกรรมแบบต่างๆ ได้ (guideline)
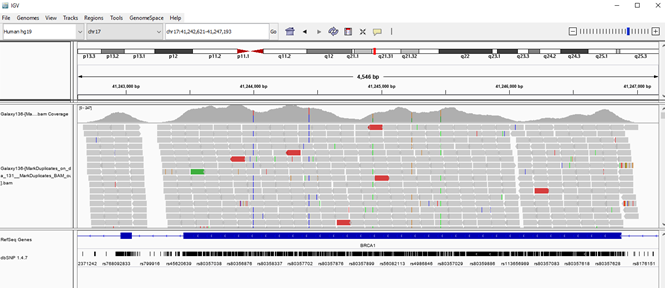
การวิเคราะห์ความผันแปรทางพันธุกรรม (variant calling)
การเปรียบเทียบ sequence read จากตัวอย่างกับจีโนมอ้างอิงจะทำให้ทราบข้อมูลความผันแปรทางพันธุกรรมที่เกิดข้นในตัวอย่างคนไข้ได้ ความผันแปรที่พบมีได้หลายแบบ ได้แก่
- SNPs ซึ่งสังเกตได้จากความแตกต่างของนิวคลีโอไทด์ในตำแหน่งนั้นเทียบกับจีโนมอ้างอิง
- small indels เป็นการขาดหายไปหรือเพิ่มเกินมาของสายดีเอ็นเอ ขนาดประมาณ 1-50 เบส
- copy number variants (CNVs) เป็นการเพิ่มจำนวนชุดของยีนหรือบริเวณในจีโนม สามารถตรวจสอบได้จากการเปลี่ยนแปลงของ coverage
- structural variants (SVs) เกิดจากการกลับทิศ (inversion) หรือเปลี่ยนตำแหน่ง (translocation) ของชิ้นส่วนดีเอ็นเอ จะสามารถตรวจสอบได้จาก sequence read ที่ไม่สามารถ map กับบริเวณของจีโนมอ้างอิงได้ต่อเนื่องทั้งเส้น (soft-clipped reads) หรือ reads ที่เมื่อ map แล้วมีขนาดของชิ้นส่วน (insert size) ทิศทาง (orientation) หรือการจัดเรียง (coordinate order) ที่ต่างจากปกติ (discordant paired-end reads)
นอกจากนี้การวิเคราะห์ความผันแปรทางพันธุกรรมในเซลล์ร่างกาย (somatic variants) กับเซลล์สืบพันธุ์ (germline variants) จะมีความแตกต่างกัน โดยการวิเคราะห์ somatic variant ต้องเปรียบเทียบกับความผันแปรในเซลล์หรือเนื้อเยื่อปกติของผู้ป่วย ในขณะที่ germline variant ต้องเปรียบเทียบกับความสมาชิกในครอบครัวเพื่อระบุ de novo variants
โปรแกรมที่ใช้สำหรับ variant calling มีหลายโปรแกรมตามลักษณะของ variants ที่สนใจ ได้แก่
- Germline callers: CRISP, GATK, Samtools, SMVer, VarScan
- Somatic callers: GATK, Samtools, SomaticSniper, VarScan
- CNV identification tools: CNVnator, RDXplorer, CONTRA, ExomeCNV
- SV identification tools: BreakDancer, Breakpointer, CLEVER, GASVPro, SVMerge
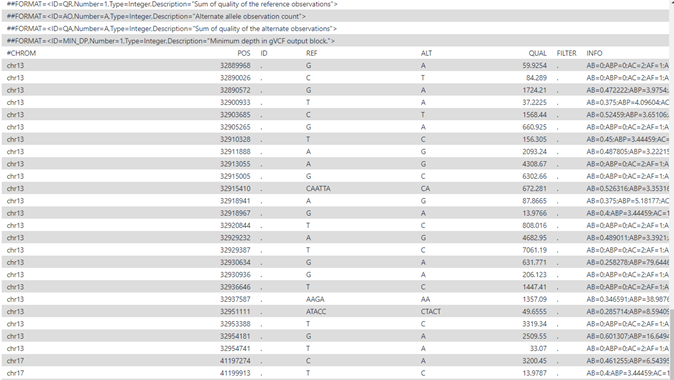
ผลของ variant calling จะบันทึกอยู่ในไฟล์ .vcf (variant calling format) ซึ่งประกอบด้วย VCF header (ข้นต้นบรรทัดด้วย ##) และส่วนของ body ที่ประกอบด้วยคอลัมหลักดังนี้
- CHROM ระบุโครโมโซม
- POS ตำแหน่งบนโครโมโซม
- ID ชื่อของยีน (identifier)
- REF รูปแบบของจีโนมอ้างอิง (reference alleles)
- ALT ความผันแปรที่พบ (alternate alleles) หากมีมากฟว่าหน่งแบบจะคั่นด้วย ;
- QUAL ค่าคุณภ่าพแสดงโอกาศที่ ALT จะผิดพลาด
- FILTER เกณฑ์การคัดเลือกตามเงื่อนไขที่กำหนดไว้
- INFO รายละเอียดเพิ่มเติม
- FORMAT ข้อมูลอื่นๆ แยกระหว่างกันด้วย ;
ขั้นตอนของการ call variant นั้นสามารถทำได้เบื้องตันโดยใช้โปรแกรม IGV (ภาพที่ 10) หรือใช้โปรแกรม Freebayes โดยไปที่หมวด Variant Calling แล้วเลือก FreeBayes โดยใช้ไฟล์ .bam และ .bai ที่ได้จากโปรแกรม Picard เป็น input และใช้ human reference genome hg19 เป็นจีโนมอ้างอิง แล้วคลิก execute
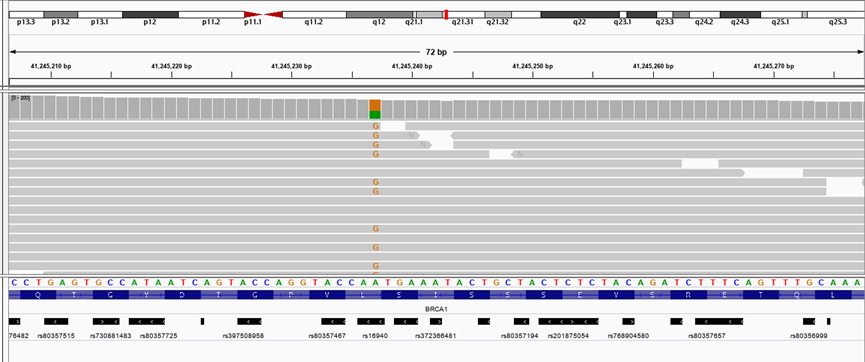
ผลที่ได้จากการ call variant ด้วยโปรแกรม FreeBayes จะอยู่ในรูปแบบไฟล์ .vcf ที่เก็บข้อมูลของ variant ตามที่อธิบายแล้วข้างต้น (ภาพที่ 11) แล้วดาวน์โหลดไฟล์ .vcf ที่ได้เพื่อเปิดแสดงด้วยโปรแกรม IGV จะปรากฎอีกหนึ่งช่องขึ้นมาในโปรแกรมและแสดงตำแหน่งของ variant (ภาพที่ 12) สามารถวิเคราะห์ตำแหน่งของ variant ชนิดของการกลาย (mutation) ว่าเป็นแบบ heterozygous (Aa) หรือ homozygous (AA และ aa) รวมถึง sequence depth ที่สนับสนุน variant ในตำแหน่งนั้น อย่างไรก็ตามข้อมูลที่ได้จากโปรแกรม call variant นี้อาจมีความผิดพลาด (artefacts) อยู่ ซึ่งอาจเกิดจาก PCR duplicates ตอนเตรียม DNA library ความผิดพลาดระหว่างกระบวนการ sequencing และความผิดพลาดจากการ mapping โดยเฉพาะในบริเวณ repeats และ low-complexity (บริเวณที่เป็น repeats สั้นๆ)
การระบุความสำคัญและหน้าที่ของ variants (variant annotation)
ขั้นตอนต่อมาภายหลังจาก variant calling คือการระบุว่า variant ที่พบในตัวอย่างนั้นมีความสำคัญต่ออาการ ความผิดปกติหรือลักษณะของผู้ป่วยอย่างไร เรียกขั้นตอนนี้ว่า variant annotation ในลำดับแรกจะต้องรู้ว่า variant นั้นพบในยีนใด (gene) ทรานส์คริปแบบใด (transcript) บริเวณใดของยีน การกลายที่พบนั้นทำให้เกิดการเปลี่ยนแปลงกับโปรตีนหรือไม่ ผลของการกลายกระทบต่อโปรตีนอย่างไร และ variant นี้พบอยู่ก่อนแล้วในฐานข้อมูล เช่น dbSNP โดยฐานข้อมูล dbSNP เก็บข้อมูลทั้ง variant จากคนปกติและผู้ป่วย
การค้นหาข้อมูลของยีนและทรานส์คริปอาจใช้ฐานข้อมูล RefSeq หรือ Ensembl ข้อมูลสำหรับการ annotation ส่วนใหญ่จะสัมพันธ์กับข้อมูล transcript และให้ข้อมูลประกอบถึงส่วนของเนื้อเยื่อที่มีการผลิต transcript รูปแบบนั้น ส่วนผลจากการกลายที่มีต่อลำดับกรดอะมิโนของโปรตีนสามารถมีได้หลายรูปแบบ ได้แก่
- Missense (non-synonymous) mutation ทำให้ชนิดของกรดอะมิโนเปลี่ยนไป
- Nonsense mutation ทำให้สายของโปรตีนสั้นลง
- Silent (synonymous) mutation ไม่เปลี่ยนแปลงชนิดของกรดอะมิโนในโปรตีน
- Insertion
- Deletion
- Duplication
- Frameshift mutation เกิดจาก indels ที่ทำให้ frame ในการอ่านรหัสพันธุกรรมเปลี่ยนไป
ลำดับต่อมาของ variant annotation คือ
- การวิเคราะห์องค์ประกอบสำคัญของโปรตีน (protein domains) ด้วยฐานข้อมูล UniProt
- การทำนายหน้าที่ของโปรตีนด้วยโปรแกรมทางชีวสารสนเทศ (in silico predictions) เพื่อศึกษาผลกระทบที่เกิดกับโปรตีน ระดับของความอนุรักษ์ (conservation) ในโปรตีนเมื่อเทียบกับต่างประชากรหรือชนิด ได้แก่ SIFT, PolyPhen, Condel, MutationTaster และ REVEL อย่างไรก็ตามการทำนายเหล่านี้จะมีน้ำหนักต่อการตัดสินน้อย มักนำไปใช้เป็นแนวทางในการยืนยันต่อด้วยการทดสอบหรือทดลอง
- ความถี่ของแอลลีลในประชากร (population frequencies) เช่น gnomAD หรือ locus specific databases โดยใช้การเปรียบเทียบความถี่ของ variant ที่สนใจกับกลุ่มประชากรที่ใกล้เคียง เพื่อเพื่อเป็นแนวทางในการพิจารณาว่า variant ที่พบนั้นปรากฏทั่วไป (common variants) หรือพบน้อย (rare variants) ในประชากร
- และค้นหาข้อมูลของ variant ในฐานข้อมูลทางการแพทย์ (clinical databases) เช่น ClinVar HGMD OMIM หรือ cancer databases เช่น COSMIC My Cancer Genome และฐานข้อมูลเฉพาะภูมิภาคหรือประเทศ เชน 100k Genomes Project
การทำ variant annotation มีหลายโปรแกรมให้เลือกใช้ ได้แก่ Annovar Variant Effect Predictor (VEP) และ CellBase
Human Genome Variation Society (HGVS) ได้กำหนดรูปแบบของ variant annotation ว่า
- จำเป็นต้องระบุรายละเอียดของ reference sequence ว่าเป็น g. = genomics c. = cDNA n. = non-coding DNA และ p. = protein
- ต้องบอกตำแหน่งของ variant (position)
- ต้องระบุ reference และ alternate allele หรือ กรดอะมิโน
การทำ variant annotation ของไฟล์ .vcf ที่ได้จากขั้น variant calling ทำได้โดยใช้โปรแกรม VEP ให้อัพโหลดไฟล์ .vcf กำหนด parameters ดังนี้
- identifiers เลือก Gene symbol Transcript version CCDS Protein และ HGVS
- Variants and frequency data เลือก 1000 Genome global minor allele frequency gnomAD (exome) allele frequencies
- Additional annotations เลือก Transcript biotype Transcript support level APPRIS MANE และ Protein domain
- Predictions เลือก SIFT และ PolyPhen
- Filtering options เลือก Return results for variants in coding regions only แล้วคลิก run (ภาพที่ 13)
เมื่อ VEP ดำเนินการวิเคราะห์เสร็จ คลิก View Results (ภาพที่ 14 และ 15) เพื่อตรวจดูผลของ variant แต่ละตำแหน่ง และสามารถดาวน์โหลดไฟล์ .vcf และ .txt เพื่อนำไปใช้รายงานผลต่อได้
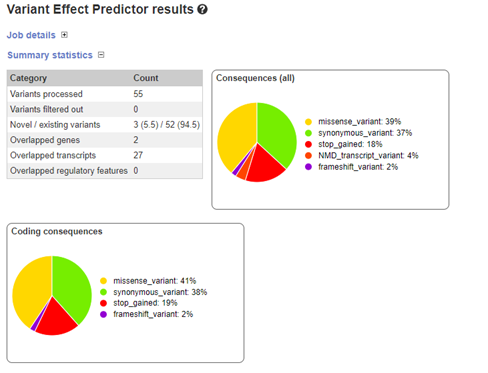
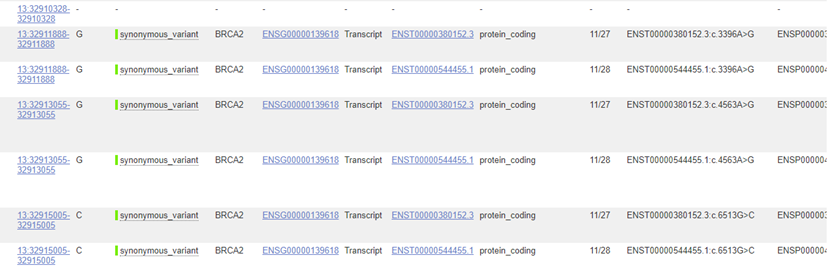
การแปลผลของ variant ในทางการแพทย์ (variant interpretation)
ข้อมูลของ variant ที่ได้จากการ annotation นั้นจำเป็นต้องผ่านกระบวนการแปลผลด้วยแพทย์ผู้ชำนาญโดยอาศัยข้อมูลและหลักฐานหลายด้านประกอบกัน เพื่อคัดเลือกหรือกลั่นกรอง (filtering) ให้ได้ variant ที่สำคัญหรือเกี่ยวข้องกับความผิดปกติของผู้ป่วยอย่างแท้จริง ได้แก่
- ข้อมูลของผู้ป่วย (patient clinical details)
- ฐานข้อมูลที่เกี่ยวข้อง (databases)
- งานวิจัยทางวิทยาศาสตร์ (scientific literature)
- โปรแกรมวิเคราะห์ทางชีวสารสนเทศ (in silico tools)
ผลกระทบของ variant อาจแบ่งเป็น 5 ระดับ (5-tier system) คือ
- 1 – Benign
- 2 – Likely Benign
- 3 – Uncertain Significance
- 4 – Likely Pathogenic
- 5 – Pathogenic
การพิจารณาผลกระทบจาก variant ต้องใช้ข้อมูลจากหลายทางประกอบกัน ได้แก่
- ข้อมูลผลกระทบของ variant (variant effect)
- ความถี่ของ variant ในคนปกติ (frequency in healthy individuals)
- ความถี่ของ variant ในคนป่วย (frequency in affected individuals)
- การศึกษาหน้าที่และการทำงานของยีนหรือโปรตีน (functional studies)
- ข้อมูลของครอบครัว (family information)
- ข้อมูล variant อื่นๆ ของผู้ป่วยคนเดียวกัน
- การทำนายด้วยโปรแกรมทางชีวสารสนเทศ
ภาพที่ 16 แสดงตัวอย่างของเกณฑ์ประกอบการพิจารณา variant

